缺失值
缺失值是现实数据集中的常见问题,处理缺失值是数据预处理的关键步骤。缺失值可能由于各种原因而发生,例如数据的结构和质量、数据输入错误、传输过程中的数据丢失或不完整的数据收集。这些缺失的值可能会影响机器学习模型的准确性和可靠性,因为它们可能会引入偏差并扭曲结果,有些模型甚至在在缺少值的情况下根本无法工作。所以在构建模型之前,适当地处理缺失值是必要的。
本文将展示如何使用三种不同级别的方法处理这些缺失值:
检查缺失的值
首先必须检查每个特性中有多少缺失值。作为探索性数据分析的一部分,我们可以使用以下代码来做到这一点:
导入pandas并在数据集中读取它们,对于下面的示例,我们将使用葡萄酒质量数据集。
importpandasaspd
df=pd.read_csv('Wine_Quality.csv')
然后可以用下面的代码行检查缺失的值。
df.isnull().sum()
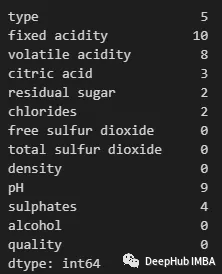
可以使用以下方法查看任何特性中包含缺失值的行:
df_filtered=df[df.isnull().any(axis=1)]
df_filtered.head()
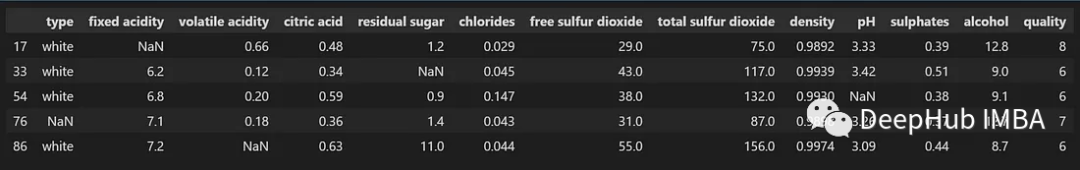
现在我们可以开始处理这些缺失的值了。
初级方法
最简单的方法是删除行或列(特性)。这通常是在缺失值的百分比非常大或缺失值对分析或结果没有显著影响时进行的。
删除缺少值的行。
df_droprows=df.dropna()
df_droprows.isnull().sum()
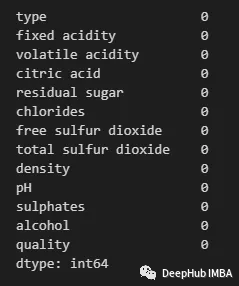
使用以下方法删除列或特性:
df_dropcols=df.drop(columns=['type', 'fixed acidity', 'citric acid', 'volatile acidity', 'residual sugar', 'chlorides', 'pH', 'sulphates'])
df_dropcols.isnull().sum()
通过删除行,我们最终得到一个更短的数据集。当删除特征时,我们最终会得到一个完整的数据集,但会丢失某些特征。
print("Shape when dropping rows: ", df_droprows.shape)
print("Shape when dropping features: ", df_dropcols.shape)

这两种方法都最直接的方法,而且都会导致丢失有价值的数据——所以一般情况下不建议使用。
均值/中值插补
下一个初级的方法是用特征的平均值或中值替换缺失的值。在这种情况下不会丢失特征或行。但是这种方法只能用于数值特征(如果使用平均值,我们应该确保数据集没有倾斜或包含重要的异常值)。
比如下面用均值来计算缺失值:
df=df.fillna(df.mean())
现在让我们检查其中一个估算值:
df[df.index==86]

如果要用中值,可以使用:
df = df.fillna(df.median())
df[df.index==86]

可以看到这里中值和平均值还是有区别的
众数
与上面的方法一样,该方法用特征的模式或最常见的值替换缺失的值。这种方法可以用于分类特征。
首先,让我们检查一下是否有一个类别占主导地位。我们可以通过value_counts方法来实现:
df['type'].value_counts()
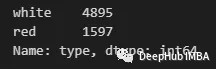
可以看到有一个“白色”数量最多。因此可以用下面的方式进行填充:
df['type'] =df['type'].fillna(df['type'].mode())
Scikit-Learn的SimpleImputer类
也可以使用Scikit-learn的SimpleImputer类执行平均值、中值和众数的插补。将策略设置为“mean”,“median”或“most_frequency”即可
df_numeric=df.drop(columns='type')
imputer_median=SimpleImputer(strategy='median')
imputer_median.fit(df_numeric)
df_imputed_median=pd.DataFrame(imputer_median.transform(df_numeric), columns=df_numeric.columns)
df_imputed_median.head()
我们也可以将策略设置为' constant ',并指定' fill_value '来填充一个常量值。
均值/中位数/众数的优点:
- 简单和快速实现
- 它保留了样本量,并降低了下游分析(如机器学习模型)的偏差风险。
- 与更复杂的方法相比,它的计算成本更低。
缺点:
- 没有说明数据的可变性或分布,可能会导致估算值不能代表真实值。
- 可能会低估或高估缺失值,特别是在具有极端值或异常值的数据集中。
- 减少方差和人为夸大相关系数在估算数据集。
- 它假设缺失的值是完全随机缺失(MCAR),这可能并不总是这样
使用领域知识进行评估
处理缺失数据的另一种可能方法是使用基于领域知识或业务规则的估计来替换缺失的值。可以通过咨询相关领域的专家,让他们提供专业的见解,这样能够估算出合理和可信的缺失值。
但是这种方法并不一定在现实中就能够很好的实施,因为我们需要专业的人士来确保它产生准确和可靠的估算,但是这样的领域专家并不多。所以我们这里把它归在初级方法中。
中级方法
还有一些稍微高级一些的威廉希尔官方网站 来填充那些缺失的值,我们将使用预测模型来解决问题。但在此之前需要更好地理解缺失值的性质。
缺失值的类型
在我们继续使用更高级的威廉希尔官方网站 之前,需要考虑一下在数据集中可能遇到的缺失类型。数据集中有不同类型的缺失,了解缺失类型有助于确定合适的方法。以下是一些常见的类型:
完全随机缺失( Missing Completely at Random):在这种类型中,缺失的值是完全随机的,这意味着一个值缺失的概率不依赖于任何观察到的或未观察到的变量。例如,如果一个受访者在调查中不小心跳过了一个问题,这就是MCAR。
随机丢失(Missing at Random):在这种类型中,一个值缺失的概率取决于观察到的变量,而不是值本身。例如,如果调查对象不太可能回答敏感问题,但不回答问题的倾向取决于可观察到的变量(如年龄、性别和教育),那么这就是MAR。
非随机丢失(Missing Not at Random):在这种类型中,一个值缺失的概率取决于未观察到的变量,包括缺失值值本身。例如,如果抑郁程度较高的个体不太可能报告他们的抑郁水平,而不报告的倾向在数据中是无法观察到的,那么这就是MNAR。
回归插补
我们将使用一个回归模型来对那些缺失的值进行有根据的猜测,通过分析数据集中的其他特征,并使用它们的相关性来填补。
在处理遵循某种模式(MAR或MCAR)的缺失数据时,回归插补特别有用。因为当特征之间存在很强的相关性时,这种方法很有效。
我们这里将创建一个不包含分类特征的数据版本。然后以为每一列的缺失值拟合线性回归模型。这里就需要使用Scikit-learn的线性回归模块。
importpandasaspd
fromsklearn.linear_modelimportLinearRegression
# Read data
df=pd.read_csv('Wine_Quality.csv')
# Make sub dataframe with only numeric features
df=df.drop(columns='type')
# Separate the columns with missing values
missing_cols=df.columns[df.isna().any()].tolist()
non_missing_cols=list(set(df.columns) -set(missing_cols))
print(missing_cols)
# loop over each column with missing values
forcolinmissing_cols:
# Create a copy of the dataframe without missing values in the current column
df_temp=df.dropna(subset=[col] +non_missing_cols)
# Split the dataframe into features (X) and target variable (y)
X=df_temp[non_missing_cols]
y=df_temp[col]
# Create and fit a linear regression model
lr=LinearRegression()
lr.fit(X, y)
# Impute missing values in the current column using the fitted model
df.loc[df[col].isna(), col] =lr.predict(df.loc[df[col].isna(), non_missing_cols])

回归插补的优点:
- 可以处理大量缺失值。
- 可以保留数据集的统计属性,例如均值、方差和相关系数。
- 可以通过减少偏差和增加样本量来提高下游分析(例如机器学习模型)的准确性。
回归插补的缺点:
- 它假设缺失变量和观察到的变量之间存在线性关系。
- 如果缺失值不是随机缺失 (MAR) 或完全随机缺失 (MCAR),则可能会引入偏差。
- 可能不适用于分类或有序变量。
- 在计算上昂贵且耗时,尤其是对于大型数据集。
(KNN) 插补
另一种方法是聚类模型,例如K-最近邻 (KNN) 来估计那些缺失值。这与回归插补类似,只是使用不同的算法来预测缺失值。
importpandasaspd
fromsklearn.imputeimportKNNImputer
# Read data
df=pd.read_csv('Wine_Quality.csv')
# Make sub dataframe with only numeric features
df=df.drop(columns='type')
# create a KNN imputer object
imputer=KNNImputer(n_neighbors=5)
# impute missing values using KNN
df=pd.DataFrame(imputer.fit_transform(df), columns=df.columns)

这里我们就要介绍一个包fancyimpute,它包含了各种插补方法:
pip install fancyimpute
使用的方法如下:
# Import the necessary libraries
importnumpyasnp
importpandasaspd
fromfancyimputeimportKNN
# Load the dataset
df=pd.read_csv('Wine_Quality.csv')
# Drop non-numeric features
df=df.drop(columns='type')
# Get list of columns with missing values
missing_cols=df.columns[df.isna().any()].tolist()
# Create an instance of the KNN imputer
imputer=KNN()
# Fit and transform the imputer to the dataset
imputed_array=imputer.fit_transform(df[missing_cols])
# Replace the missing values in the original dataset
df[missing_cols] =imputed_array
# View the imputed dataset
df
KNN 插补的优点:
- 可以捕获变量之间复杂的非线性关系。
- 不对数据的分布或变量之间的相关性做出假设。
- 比简单的插补方法(例如均值或中值插补)更准确,尤其是对于中小型数据集。
缺点:
- 计算上可能很昂贵,尤其是对于大型数据集或高维数据。
- 可能对距离度量的选择和选择的最近邻居的数量敏感,这会影响准确性。
- 对于高度倾斜或稀疏的数据表现不佳。
高级方法
通过链式方程 (MICE) 进行多元插补
MICE 是一种常用的估算缺失数据的方法。它的工作原理是将每个缺失值替换为一组基于模型的合理值,该模型考虑了数据集中变量之间的关系。
该算法首先根据其他完整的变量为数据集中的每个变量创建一个预测模型。然后使用相应的预测模型估算每个变量的缺失值。这个过程重复多次,每一轮插补都使用前一轮的插补值,就好像它们是真的一样,直到达到收敛为止。
然后将多个估算数据集组合起来创建一个最终数据集,其中包含所有缺失数据的估算值。MICE 是一种强大而灵活的方法,可以处理具有许多缺失值和变量之间复杂关系的数据集。它已成为许多领域(包括社会科学、健康研究和环境科学)中填补缺失数据的流行选择。
fancyimpute包就包含了这个方法的实现,我们可以直接拿来使用
importnumpyasnp
importpandasaspd
fromfancyimputeimportIterativeImputer
# Read data
df=pd.read_csv('Wine_Quality.csv')
# Convert type column to category (so that miceforest can handle as a categorical attribute rather than string)
df=df.drop(columns='type')
# Get list of columns with missing values
missing_cols=df.columns[df.isna().any()].tolist()
# Create an instance of the MICE algorithm
imputer=IterativeImputer()
# Fit the imputer to the dataset
imputed_array=imputer.fit_transform(df[missing_cols])
# Replace the missing values in the original dataset
df[missing_cols] =imputed_array
# View the imputed dataset
df
这个实现没法对分类变量进行填充,那么对于分类变量怎么办呢?
MICEforest
MICEforest 是 MICE的变体,它使用 lightGBM 算法来插补数据集中的缺失值,这是一个很奇特的想法,对吧。
我们可以使用 miceforest 包来实现它
pipinstallmiceforest
#或
condainstall-cconda-forgemiceforest
使用也很简单:
importpandasaspd
importmiceforestasmf
# Read data
df=pd.read_csv('Wine_Quality.csv')
# Convert type column to category (so that miceforest can handle as a categorical attribute rather than string)
df['type'] =df['type'].astype('category')
# Create an instance of the MICE algorithm
imputer=mf.ImputationKernel(data=df,
save_all_iterations=True,
random_state=42)
# Fit the imputer to the dataset. Set number of iterations to 3
imputer.mice(3, verbose=True)
# Generate the imputed dataset
imputed_df=imputer.complete_data()
# View the imputed dataset
imputed_df
可以看到,分类变量 'type' 的缺失值已经被填充了
总结
我们这里介绍了三个层级的缺失值的处理方法,这三种方法的选择将取决于数据集、缺失数据的数量和分析目标。也需要仔细考虑输入缺失数据对最终结果的潜在影响。处理缺失数据是数据分析中的关键步骤,使用合适的填充方法可以帮助我们解锁隐藏在数据中的见解,而从主题专家那里寻求输入并评估输入数据的质量有助于确保后续分析的有效性。
-
机器学习
+关注
关注
66文章
8414浏览量
132606 -
python
+关注
关注
56文章
4795浏览量
84658 -
Mar
+关注
关注
0文章
4浏览量
5304
发布评论请先 登录
相关推荐
STM32H743ADC数据转换输出值缺失的原因?
如何实现VISA三个不同延迟的独立循环抓值
处理数据缺失的结构化解决办法
FPGA三个按键给同一个信号赋三个不同的值按键回弹为 000 后变量的值将改变怎么解决?
无线传感网络缺失值估计方法
工业4.0的工厂自动化系统通常主要包括三个层级的设备




 处理缺失值的三个层级的方法总结
处理缺失值的三个层级的方法总结











评论